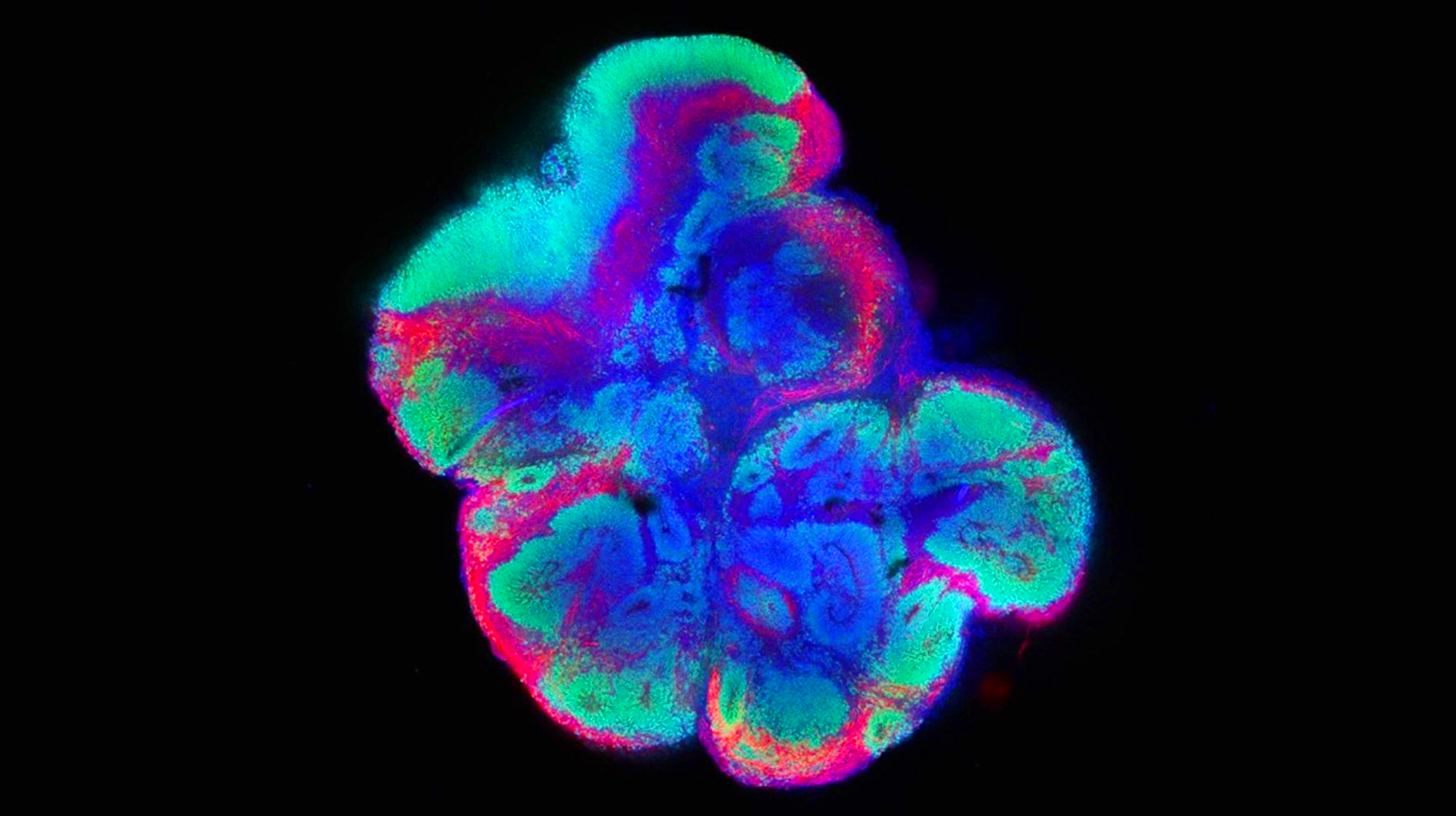
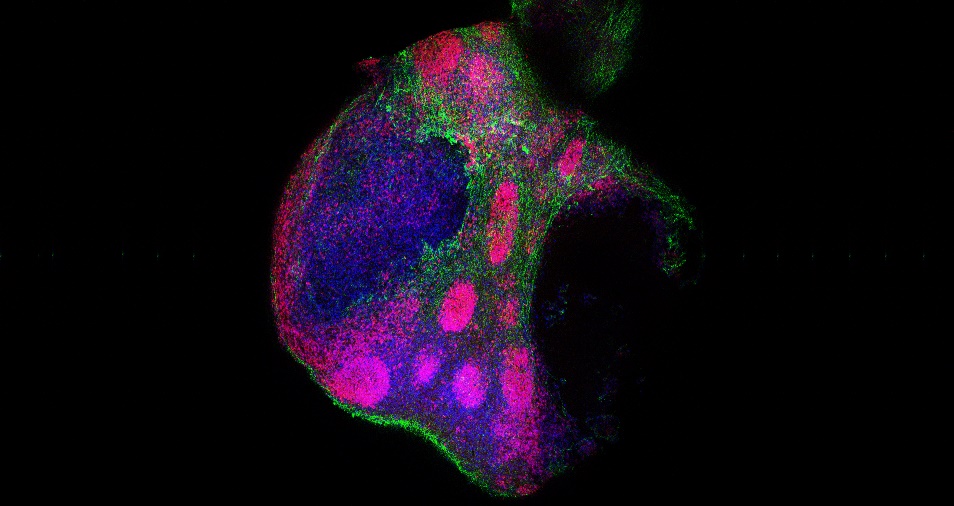
La bioinformática, clave en la biomedicina moderna y en la actual pandemia
A paragraph is a self-contained unit of a discourse in writing dealing with
a particular point or idea. Paragraphs are usually an expected part of
formal writing, used to organize longer prose.